Desde 2012, el sistema CRISPR-Cas se ha convertido en la herramienta de edición genómica favorita en los laboratorios de biología molecular. Esto se debe a su precisión y facilidad de uso para editar células, tejidos y organismos completos. Por ello, el uso de la tecnología CRISPR-Cas está muy extendido en la investigación experimental y aplicada.
En los últimos años se han desarrollado potentes herramientas para la manipulación del genoma en animales, plantas y microorganismos basadas en CRISPR-Cas9. Ya hemos hablado sobre qué es y cómo funciona la tecnología CRISPR en anteriores publicaciones: las 5 preguntas clave para la tecnología CRISPR, cómo evitar los efectos off-target en un experimento CRISPR o cómo diseñar guías para CRISPR.
Aunque cada día surgen nuevos usos, en este post queremos resaltar algunas de las principales aplicaciones CRISPR.
En Abyntek ponemos a tu disposición los productos CRISPR que necesitas para tu investigación y te asesoramos acompañándote durante todo el proceso.
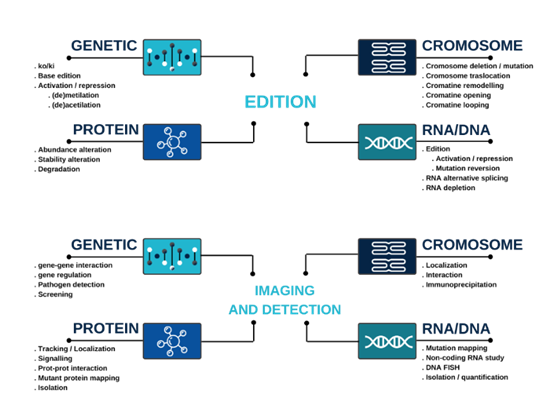
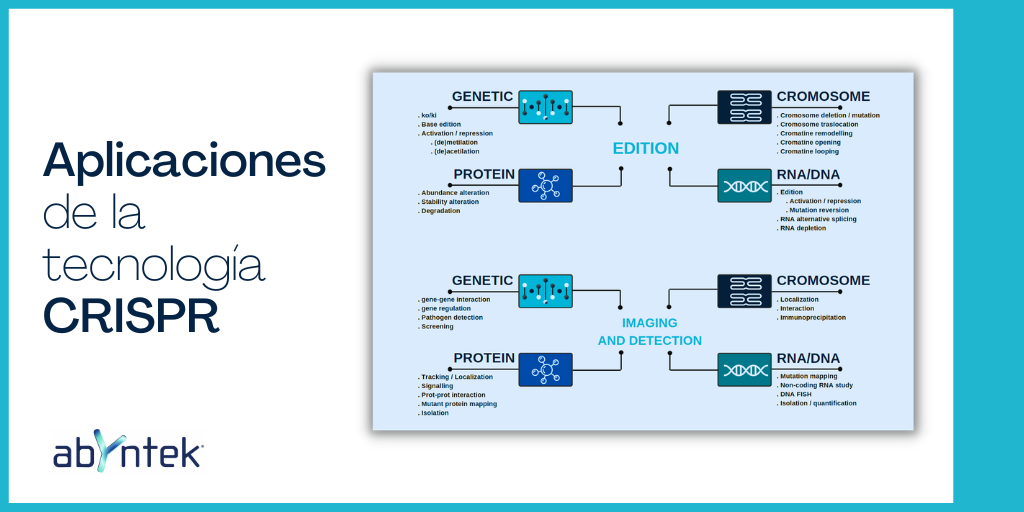
Edición
Mutación inducida
El sistema CRISPR-Cas9 es conocido mundialmente como una herramienta de edición del genoma. Cas9 corta el ADN de doble cadena para generar una ruptura que será reparada por la maquinaria de reparación celular.
Ya hemos comentado previamente que existen 2 mecanismos de reparación, ambos generando introducción de mutaciones, unión de extremos no homólogos (NHEJ) o reparación dirigida por homología (HDR). NHEJ produce pequeñas inserciones o deleciones en el sitio de escisión, denominadas indels, mientras que HDR usa una plantilla de ADN para reemplazar mediante recombinación la secuencia diana con una secuencia alternativa.
Existen otras vías de reparación del ADN, como el “annelling” de una sola hebra, la unión de extremos alternativos, la unión mediada por microhomología, el desajuste y la reparación por escisión de bases y nucleótidos.
Regulación
El sistema CRISPR-Cas tiene el potencial de usarse para regular la expresión génica endógena, incluida la regulación tanto transcripcional como epigenética.
Para ello, se han desarrollado variantes desactivadas de Cas9 introduciendo dos mutaciones puntuales. Estas mutaciones desactivan la actividad de rotura del ADN de Cas9, pero no afectan a la de unión. Esta nueva Cas9 inactiva se denominó Cas9 muerta (dCas9).
Posteriormente, si se fusionan activadores o represores transcripcionales a la dCas9, el sistema CRISPR-dCas9 puede usarse para activar o inhibir la transcripción de genes de interés. Además, también permite la manipulación simultánea de múltiples genes dentro de una célula.
Sistemas de edición de bases
Uno de los últimos usos de CRISPR es la edición de bases, que permite sustituciones precisas de nucleótidos de manera programable, sin producir un DBS y sin requerir un patrón o “template”.
Los editores de bases de ADN están formados por 2 componentes, una proteína Cas y una enzima modificadora de ADN monocatenario. Se han descrito dos clases de editores de bases de ADN (BE): editores de bases de citosina (CBE) y editores de bases de adenina (ABE). Son capaces de introducir mutaciones de transición C→T, T→C, A→G y G→A.
Sin embargo, los editores de bases tienen una limitación importante: no pueden generar ediciones precisas más allá de las cuatro mutaciones de transición mencionadas. Para superar estas deficiencias, se desarrollaron herramientas de edición “prime”, que pueden generar inserciones, eliminaciones y los 12 tipos de sustituciones de bases.
Los editores “prime” utilizan una transcriptasa inversa (RT) modificada fusionada con la nickasa Cas9 y un ARN guía de edición principal (pegRNA). El pegRNA contiene no solo la secuencia complementaria a la diana que dirige nCas9 a su secuencia objetivo, sino también una secuencia adicional que determina los cambios de secuencia deseados. Después de que nCas9 corta el sitio de destino, la transcriptasa inversa utiliza el pegRNA como plantilla para la transcripción inversa y, en consecuencia, modifica la región de destino de manera programable.
Sistemas de edición de ARN
Además de editar el ADN, los sistemas CRISPR-Cas también pueden editar ARN.
El sistema CRISPR-Cas13 contiene en una proteína Cas13 guiada por una secuencia de ARN de cadena simple, con actividad de ribonucleasa. De esta manera, puede unirse al ARN monocatenario diana (ssRNA) y generar una rotura específica.
Basados en Cas13, se han desarrollado 2 sistemas de edición de ARN, fusionando Cas13 catalíticamente inactivada (dCas13) con el dominio adenina/citidina desaminasa de ADAR2 (adenosina desaminasa que actúa sobre el ARN tipo 2). Se trata del sistema REPAIR, que permite la sustitución A-Gm, y el sistema RESCUE, que permite el reemplazo de C-U.
En comparación con la edición de ADN, la edición de ARN es altamente eficiente y específica. Además, puede realizar ediciones genéticas temporales y por tanto reversibles.
Imagen y detección
Además de las aplicaciones de edición y regulación del genoma, uno de los últimos diseños de la tecnología CRISPR/Cas ha permitido utilizar este sistema para la visualización de elementos genómicos en células vivas.
Escaneo en vivo de ADN/ARN mediante CRISPR/Cas
La visualización del ADN es una aplicación importante para comprender una variedad de procesos celulares, como la replicación, la transcripción y la recombinación, y las interacciones entre el ADN y las proteínas asociadas y el ARN.
dCas9 también permite la fusión de fluoróforos. Esto permite la visualización de secuencias específicas de ADN y una observación dinámica de la organización de la cromatina a lo largo del ciclo celular.
Además de la obtención de imágenes de ADN, el sistema CRISPR/Cas9 también se puede utilizar para la obtención de imágenes de ARN. Las modificaciones a la secuencia de gRNA permiten el reconocimiento y seguimiento del mRNA.
Estas nuevas aplicaciones mejoran las metodologías existentes para la obtención de imágenes en vivo dentro de las células, como la hibridación in situ con fluorescencia (FISH) y el marcado fluorescente de proteínas de unión al ADN. FISH utiliza sondas de ácido nucleico marcadas con fluorescencia para unir y visualizar el ADN, resultando el método CasFISH más simple, menos costoso y más rápido.
CRISPR/Cas9-mediated Chromatin Immunoprecipitation
La inmunoprecipitación de cromatina (CHiP) es un método utilizado para la caracterización de proteínas y ARN asociados a la cromatina. Las modificaciones al sistema CRISPR/Cas9 permiten el aislamiento de estas regiones genómicas. Se puede introducir una señal de localización nuclear y una etiqueta de epítopo en dCas9 para crear una proteína de unión al ADN que puede ser el objetivo de los ARN guía de CRISPR.
El complejo de cromatina CRISPR/Cas9 se puede purificar siguiendo las técnicas tradicionales de inmunoprecipitación de cromatina (CHiP), en las que utilizan anticuerpos. Este método adaptado solo necesita un único anticuerpo contra la proteína Cas9 para purificaciones a gran escala, mientras que CHiP tradicional requiere el uso de múltiples anticuerpos contra cada proteínas.
DNA/RNA detection
El reconocimiento específico y rápido ácidos nucleicos basado en la tecnología CRISPR/Cas es posible. Esta técnica combina la preamplificación de ácidos nucleicos con la enzimología CRISPR-Cas.
Se basan en otras enzimas Cas, diferentes de Cas9. Estos son, Cas13 y Cas12. El que usa Cas13 se conoce comúnmente como SHERLOCK (Specific High Sensitivity Enzymatic Reporter UnLOCK- ing) y está compuesto por Cas13a, un sgRNA dirigido a secuencias específicas de RNA e indicadores de RNA fluorescentes. Después de que la proteína Cas13a reconozca y escinda el ARN diana, cortará el indicador de ARN y liberará la señal de fluorescencia detectable, identificando así la secuencia diana. Este método se utiliza para detectar virus o bacterias patógenas.
Además de Cas13, Doudna et al. desarrollaron un sistema de detección de ácidos nucleicos basado en la enzima Cas12a, llamado DETECTR (DNA endonuclease-targeted CRISPR trans-reporter). Este método depende de la actividad colateral de la proteína Cas12a que después de reconocer el ARN diana, degrada todas las moléculas de ADN adyacentes. La degradación del ADN libera fluoróforos y da como resultado una señal fluorescente fuerte y estable detectada por un fluorímetro. Además, DETECTR se ha combinado con un paso de preamplificación isotérmica para una mayor detección de las secuencias objetivo.
Las herramientas de diagnóstico SHERLOCK y DETECTR son sondas altamente sensibles y específicas en comparación con los métodos tradicionales basados en PCR. Además, no requieren equipos sofisticados y tienen un costo estimado menor.
Screening
El sistema CRISPR-Cas9 permite la detección de alto rendimiento en todo el genoma (Genome-wide high- throughput screening).
El cribado CRISPR es un enfoque experimental a gran escala que facilita el descubrimiento de secuencias genéticas relacionadas con funciones o fenotipos específicos. Se nutre de bibliotecas con una gran cantidad de sgRNAs, utilizados para identificar genes relacionados con un fenotipo particular. Este tipo de selección podría usarse no solo para genes funcionales y esenciales, sino también para secuencias no codificantes.
En la actualidad, se han construido bibliotecas de sgRNA que comprenden todo el genoma humano y de ratón, y mejorándose cada vez más.
En Abyntek contamos con un extenso catálogo y un servicio a medida con productos validados por el Broad Institute MIT y Harvard donde nuestros expertos te asesorarán y guiarán en todo momento para impulsar tu investigación a otro nivel.
Contáctanos a través de este formulario o enviando un email a info@abyntek.com. ¡Estaremos encantados de ayudarte!
Bibliografía
Barrangou R, Doudna JA. Applications of CRISPR technologies in research and beyond. Nat Biotechnol. 2016;34(9):933-941. doi: 10.1038/nbt.3659. Epub 2016 Sep 8. PMID: 27606440.
Xu Y, Li Z. CRISPR-Cas systems: Overview, innovations and applications in human disease research and gene therapy. Comput Struct Biotechnol J. 2020 Sep 8;18:2401-2415. doi: 10.1016/j.csbj.2020.08.031. PMID: 33005303; PMCID: PMC7508700.
Deng, Wulan, et al. «CASFISH: CRISPR/Cas9-mediated in situ labeling of genomic loci in fixed cells.» Proceedings of the National Academy of Sciences 112.38 (2015): 11870-11875.
Kantor A, McClements ME, MacLaren RE. CRISPR-Cas9 DNA Base-Editing and Prime-Editing. Int J Mol Sci. 2020;21(17):6240. Published 2020 Aug 28. doi:10.3390/ijms21176240