Cada vez son más las investigaciones que se centran en el estudio de la microbiota gastrointestinal como causa o potencial tratamiento de diversas patologías. Para ahondar en los últimos avances en este campo, en esta entrada os traemos una revisión de la aplicación de los trasplantes fecales para el tratamiento de infecciones recurrentes por Clostridium difficile, las implicaciones de las variantes genéticas en la composición del microbioma intestinal y los modelos para establecer relación de causalidad entre las alteraciones de la microbiota intestinal y determinadas patologías.
Trasplante fecal para tratar infecciones por clostridium difficile
La infección por C.difficile es la principal causa de diarrea infecciosa que se observa en el ámbito hospitalario. Aunque algunos pacientes pueden permanecer asintomáticos tras la exposición a este patógeno, muchos otros desarrollan síntomas que pueden ir desde una leve diarrea hasta el desarrollo de colitis y su consecuente riesgo mortal. Uno de los principales factores de riesgo para contraer la enfermedad es la toma de antibióticos, dado que ésta conlleva una alteración en la barrera intestinal. Los antibióticos, que tan necesarios son para tratar enfermedades potencialmente mortales, también ejercen su acción en nuestra microbiota intestinal, alterando su función protectora y facilitando el crecimiento de C.difficile. La edad avanzada (>60 años) así como presentar otras comorbilidades son también factores de riesgo que aumentan la tasa de mortalidad.
Para hacer frente a esta infección se emplean agentes microbianos específicos contra C.difficile. Sin embargo, la eficacia de estos tratamientos es limitada debido a que no actúan sobre el principal mecanismo de infección: las esporas. Éstas pueden encontrarse en el ambiente e incluso entrar en la cadena alimentaria. Una estrategia que ha permitido tratar de forma eficaz las infecciones recurrentes de C.difficile es el trasplante de microbiota fecal (FMT, por sus siglas en inglés). Este procedimiento consiste en aplicar una solución fecal de un donante sano en el tracto intestinal del paciente, con el objetivo de reestablecer la composición de su microbiota intestinal y dificultar la propagación del patógeno. El éxito obtenido mediante esta estrategia pone de manifiesto la estrecha relación que existe entre la composición de nuestra microbiota intestinal y la susceptibilidad a la infección por C.difficile.
¿INFLUYE LA GENÉTICA EN LA COMPOSICIÓN DE LA MICROBIOTA INTESTINAL?
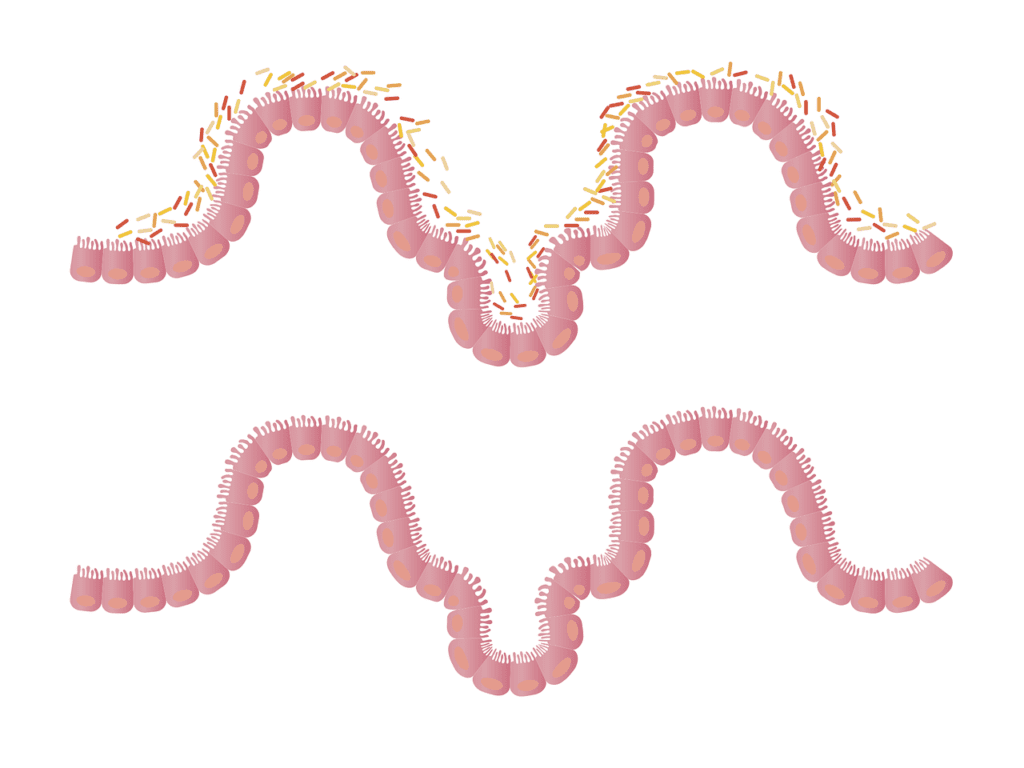
Cabe destacar, que el papel que juega la microbiota gastrointestinal va más allá de la infección por C.difficile. Numerosas patologías humanas se han asociado con una microbiota fecal que es, en determinados aspectos, distinta a la microbiota de individuos sanos. Esto es, se observan diferencias en la diversidad y abundancia relativa de determinadas especies microbianas, así como en los metabolitos producidos por dichas especies. El término ´disbiosis´ hace referencia a estas alteraciones en la microbiota que se salen de lo ´normal´ y se han observado en pacientes con alergias, Parkinson o enfermedad intestinal inflamatoria, entre otras.
Ahora bien, ¿qué es lo ”normal”? Pese a que el campo de la microbiota avanza a pasos agigantados, aún no se ha definido en profundidad qué características tiene una microbiota ”sana” o ”normal“. Numerosos factores como la dieta, la toma de antibióticos, la edad o, incluso, factores medioambientales afectan a la composición de la microbiota gastrointestinal a nivel individual. Es más, esta variabilidad está también determinada genéticamente. Varios estudios han demostrado en los últimos años cómo algunas variantes genéticas o polimorfismos de un solo nucleótido (SNPs), se asocian con una determinada composición de la microbiota gastrointestinal. Sin embargo, a día de hoy es difícil establecer conclusiones sólidas respecto a estas asociaciones debido a que, en la mayoría de los casos, los SNPs identificados varían de un estudio a otro. Existe, sin embargo, una excepción sobre la que sí parece haber consenso en los diferentes estudios:
Variantes en el gen de la enzima lactasa (LCT) se asocian significativamente con una mayor abundancia relativa de Bifidobacterium en el tracto intestinal. En particular, la presencia del genotipo CC en el SNP rs4988235, se relaciona con una baja actividad enzimática. Como Bifidobacterium obtiene energía preferentemente metabolizando la lactosa, se podría especular que, los individuos con una alta actividad enzimática tienen menor abundancia de Bifidobacterium en su microbiota, puesto que la disponibilidad de lactosa es menor. Y al contrario: aquellos individuos con una menor actividad enzimática, dispondrán de más lactosa propiciando así el crecimiento de Bifidobacterium.
No obstante, es probable que las asociaciones genéticas observadas, estén en menor o mayor medida influenciadas por factores ambientales. En particular, el consumo de productos con lactosa podría estar influenciando la asociación observada para el gen LCT. Por este motivo, estudiar los efectos de ambos factores, genética y ambiente, será imprescindible para establecer conclusiones consistentes.
ESTABLECER RELACIÓN CAUSAL ENTRE DISBIOSIS INTESTINAL Y PATOLOGÍA
Por otro lado, otra cuestión aún por establecer es la naturaleza de la relación existente entre disbiosis intestinal y patología. ¿Son los cambios en la microbiota responsables de ciertas enfermedades o son consecuencia de las mismas? Establecer una relación causa-efecto es esencial si se quiere considerar la microbiota gastrointestinal como una diana terapéutica válida para el tratamiento de diversas patologías.

Para determinar dicha relación causal el modelo más utilizado consiste en realizar un trasplante de microbiota fecal procedente de individuos con o sin enfermedad a ratones de experimentación que no poseen microbiota (germ-free mice) y realizar un análisis comparativo entre ambos grupos monitoreando el desarrollo de la enfermedad. Este modelo, conocido como Human Microbiota-Associated mice (HMA mice), no sólo permite establecer una relación causal, sino también investigar el mecanismo molecular subyacente al desarrollo de la enfermedad y evaluar posibles intervenciones terapéuticas.
A pesar de que tiene ciertas limitaciones- la extrapolación de los resultados de ratón a humano debe de hacerse de forma cautelosa- este modelo ha servido para demostrar que Helicobacter pylori es uno de los principales causantes de úlceras pépticas y que la microbiota tiene un papel protector frente a C.difficile. No obstante, aún no existen estudios robustos y concluyentes que demuestren una relación causal entre la microbiota intestinal y otras patologías, como la diabetes tipo 2, la artritis reumatoide o enfermedades neurológicas, aunque sí hay claros estudios que correlacionan estas enfermedades con una disbiosis gastrointestinal. Las investigaciones futuras que arrojen luz sobre esta cuestión estableciendo la presencia o ausencia de causalidad serán cruciales para sentar las bases de una nueva era de terapias destinadas a modular nuestra microbiota para tratar enfermedades.
REFERENCIAS:
[1] Blekhman, Ran, Julia K Goodrich, Katherine Huang, Qi Sun, Robert Bukowski, Jordana T Bell, Timothy D Spector, et al. 2015. “Host Genetic Variation Impacts Microbiome Composition across Human Body Sites.” Genome Biology 16 (1): 191. https://doi.org/10.1186/s13059-015-0759-1.
[2] Cahana, Inbal, and Fuad A Iraqi. 2020. “Impact of Host Genetics on Gut Microbiome: Take-Home Lessons from Human and Mouse Studies.” Animal Models and Experimental Medicine 3 (3): 229–36. https://doi.org/10.1002/ame2.12134.
[3] Durack, Juliana, and Susan V Lynch. 2019. “The Gut Microbiome: Relationships with Disease and Opportunities for Therapy.” The Journal of Experimental Medicine 216 (1): 20–40. https://doi.org/10.1084/jem.20180448.
[4] Turpin, Williams, Osvaldo Espin-Garcia, Wei Xu, Mark S Silverberg, David Kevans, Michelle I Smith, David S Guttman, et al. 2016. “Association of Host Genome with Intestinal Microbial Composition in a Large Healthy Cohort.” Nature Genetics 48 (11): 1413–17. https://doi.org/10.1038/ng.3693.
[5] Walter, Jens, Anissa M Armet, B Brett Finlay, and Fergus Shanahan. 2020. “Establishing or Exaggerating Causality for the Gut Microbiome: Lessons from Human Microbiota-Associated Rodents.” Cell 180 (2): 221–32. https://doi.org/10.1016/j.cell.2019.12.025.
Si está interesado en recolectar muestras fecales para su propio estudio de la microbiota, obtenga aquí más información sobre los kits OMNI-Gene GUT.
Descubra aquí también los kits de recolección de ADN a partir de saliva (Oragene DNA) para llevar a cabo estudios genéticos.